
Die Typisierung von Bakterien zur Identifizierung von verbreiteten Isolaten mit gleichem genetischem Fingerabdruck ist für die epidemiologische Überwachung von großer Bedeutung. Für Mycobacterium tuberculosis (dem Erreger der Tuberkulose, TB) und ähnlichen Erregern ist die MIRU-VNTR Typisierung weit verbreitet. Die Methode erlaubt auch die Zuordnung der Erreger in Untergruppen (Lineages).
Zusammen mit führenden Wissenschaftlern im Bereich der MIRU-VNTR-Typisierung habe ich am Universitätsklinikum Münster in den Jahren 2006-2008 eine Webanwendung entwickelt, welche die Analyse von MIRU-VNTR Daten und die Zuordnung von Isolaten zu Lineages ermöglicht. Die Planung und Umsetzung der Webanwendung wurde von mir im Rahmen meiner Dissertation selbständig durchgeführt.
Die Anwendung ist unter
 http://www.miru-vntrplus.org erreichbar und wird weltweit verwendet.
http://www.miru-vntrplus.org erreichbar und wird weltweit verwendet.
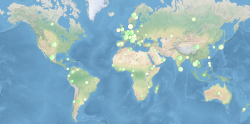 Nutzerverteilung erstes Halbjahr 2014. Die Anzahl der Nutzer pro Land ist durch die Kreisgröße dargestellt, die Anzahl der analysierten Datensätze durch die Farbe. |
Technik
Die Umsetzung erfolgte mittels Java und JavaServer Faces (JSF, Apache MyFaces). Wo es sinnvoll ist, kommen Servlets und JavaServer Pages (JSP) zum Einsatz. Zur intuitiven Gestaltung der Benutzeroberfläche werden AJAX-Komponenten für JSF (JBoss RichFaces 3) auch JavaScript verwendet.Zur Speicherung der Typisierungsdaten werden aufgrund der geringen Datenmenge und der sehr seltenen Änderungen CSV-Dateien verwendet. Eine Nomenklatur-Komponente wurde mit MySQL und Hibernate umgesetzt. Die Nomenklatur kann u.a. über eine SOAP-Schnittstelle abgefragt werden, die mit Apache Axis2 realisiert wurde.
Veröffentlichungen
Weniger T, Krawczyk J, Supply P, Harmsen D, Niemann S. Online tools for polyphasic analysis of Mycobacterium tuberculosis complex genotyping data: now and next. Infect Genet Evol 2012 Jun;12(4):748-54 [Pubmed]Weniger T, Krawczyk J, Supply P, Niemann S, Harmsen, D. MIRU-VNTRplus: a web tool for polyphasic genotyping of Mycobacterium tuberculosis complex bacteria. Nucleic Acids Res 2010, 38 Suppl:W326-331 [Pubmed],
 [PDF]
[PDF]
Allix-Béguec C, Harmsen D, Weniger T, Supply P, Niemann S. Evaluation and user-strategy of MIRU-VNTRplus, a multifunctional database for online analysis of genotyping data and phylogenetic identification of Mycobacterium tuberculosis complex isolates. J Clin Microbiol 2008, 46(8):2692-2699 [Pubmed]
Eine Liste der Veröffentlichungen, die MIRU-VNTRplus verwenden ist bei
 Google Scholar zu finden.
Google Scholar zu finden.
Die Anwendung ist Teil
 meiner Dissertation, die hier zu finden ist (PDF, 3.2 MB).
meiner Dissertation, die hier zu finden ist (PDF, 3.2 MB).
Screenshots
Einige Screenshots. Zum Vergrößern bitte anklicken.
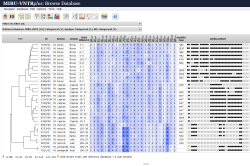 Hauptseite mit aktiviertem phylogenetischen Baum |
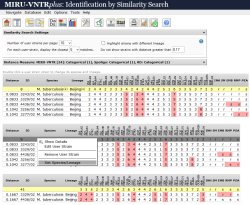 Identifizierung von Lineages basierend auf MIRU-VNTR Daten |
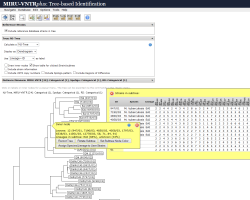 Identifizierung von Lineages mit Hilfe eines phylogenetischen Baumes |
|